グリコーゲンホスホリラーゼ
| グリコーゲンホスホリラーゼ | |||||||||
|---|---|---|---|---|---|---|---|---|---|
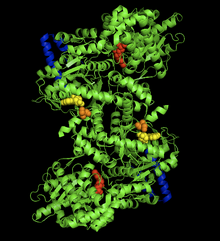 ウサギの筋のグリコーゲンホスホリラーゼ-AMP複合体の結晶構造。AMP アロステリック部位(黄色)、リン酸化したSer14(橙色)、グリコーゲン結合部位(青色)、触媒部位(赤色)。[1] | |||||||||
| 識別子 | |||||||||
| EC番号 | 2.4.1.1 | ||||||||
| CAS登録番号 | 9035-74-9 | ||||||||
| データベース | |||||||||
| IntEnz | IntEnz view | ||||||||
| BRENDA | BRENDA entry | ||||||||
| ExPASy | NiceZyme view | ||||||||
| KEGG | KEGG entry | ||||||||
| MetaCyc | metabolic pathway | ||||||||
| PRIAM | profile | ||||||||
| PDB構造 | RCSB PDB PDBj PDBe PDBsum | ||||||||
| 遺伝子オントロジー | AmiGO / QuickGO | ||||||||
| |||||||||
グリコーゲンホスホリラーゼ(EC 2.4.1.1はホスホリラーゼという酵素の一種である)。グリコーゲンホスホリラーゼは、動物におけるグリコーゲン分解の律速段階を触媒し、末端のα-1,4-グリコシド結合を切ってグルコース-1-リン酸を遊離させる。グリコーゲンホスホリラーゼは可逆的リン酸化とアロステリックな効果の両方のモデル酵素としても研究されている。
機構
[編集]反応全体は次のように示される。
グリコーゲン(n残基) + Pi ↔ グリコーゲン(n-1残基) + D-グルコース-1-リン酸[2]
グリコーゲンホスホリラーゼはグリコーゲンをグルコース単位に分解する。 グリコーゲンはグルコースが一分子少なくなり、遊離するグルコース分子は グルコース-1-リン酸となる。 代謝されるには、ホスホグルコムターゼによってグルコース-6-リン酸に変換される必要がある。
この反応は溶液中では可逆的だが、細胞内では上で示した方向にしか進まない。それは、細胞内では無機リン酸の濃度がグルコース-1-リン酸の濃度よりはるかに高いからである。[2]
グリコーゲンホスホリラーゼは直鎖状のグリコーゲン(α1-4グリコシル結合)にしか作用しない。その作用はα1-6 分枝(グリコーゲンでは非常に多く存在する)から4残基のところで止まってしまう。その状況では、グリコーゲン鎖を直鎖状にするグリコーゲン脱分枝酵素が必要である。その転移酵素は3つのグリコシル残基をまとめてその他の末端に移し、α1-6グリコシダーゼが新しくできた鎖に残ったα1-6グルコース1残基を分解する。これが完了すると、グリコーゲンホスホリラーゼが反応を続ける。この酵素はα1-4結合特異的であり、その分子には30Åの長さの割れ目があり、グリコーゲン鎖のヘリックスの半径と同じである。そこには4-5グリコシル残基が結合するが、分枝には狭すぎる。この割れ目はグリコーゲン貯蔵部位と活性を持つ触媒部位をつなげる。
グリコーゲンホスホリラーゼはピリドキサールリン酸(PLP、ビタミンB6より派生)を各触媒部位にもつ。ピリドキサールリン酸は塩基性の残基(この場合Lys680)と結合し、共有結合的にシッフ塩基を形成する。このシッフ塩基が形成されるとPLP分子が活性部位に保持され、PLPのリン酸基が無機リン酸を容易にプロトン化し、次いでα-1,4グリコシド結合を形成する酸素によって脱プロトン化されるようになる。PLPはその負の電荷がリン酸基内のみならず、ピリジン環によっても安定化されるので、容易に脱プロトン化し、そのPLPの脱プロトン化によって生じた共役塩基は非常に安定である。プロトン化した酸素は良い遊離基であり、SN1反応様式で末端のグリコーゲンからグリコーゲン鎖が離れ、その結果1位に2級カルボカチオンをもつグルコースが生じる。最後に脱プロトン化した無機リン酸が求核剤として働いてカルボカチオンと結合し、グルコース-1-リン酸とグルコース1分子分短くなったグリコーゲン鎖が生じる。
半イス型立体配座での正に荷電した酸素が関わるもう一つの機構も提唱されている。[3]
構造
[編集]グリコーゲンホスホリラーゼの単量体は大きなタンパク質であり、筋細胞では842アミノ酸からなり、分子質量は97.434 kDaである。その酵素は不活性な単量体か生物学的に活性のある2量体(2つの同一サブユニット)で存在する。[4]

グリコーゲンホスホリラーゼ2量体は、触媒部位、グリコーゲン結合部位、アロステリック部位、および可逆的にリン酸化されるセリン残基を含む、生物学的に重要な多くの領域を持つ。まず、触媒部位は比較的隠されていて、タンパク質の表面とサブユニットの境界から15Åはなれている。[5] このように表面から触媒部位へは接触が難しいので、タンパク質の活性が調節によって非常に影響を受ける。小さなアロステリックな影響がグリコーゲンの活性部位への接触しやすさを大きく増加させることがある。
おそらく最も重要な調節部位はSer14であり、それはサブユニット境界に非常に近く、可逆的なリン酸化を受ける部位である。リン酸化による構造変化は、ホスホリラーゼbをホスホリラーゼaに変換し、もともとは不規則な配列の10番目から22番目の残基をαヘリックスにする。この変化はAMPが無くともホスホリラーゼ活性を25%増加し、AMPによる活性化をさらに高める。[6]
グリコーゲンホスホリラーゼの筋でのアイソフォームで結合するAMPのアロステリック部位は、ちょうどSer14のようにサブユニット境界に近い。この部位へのAMPの結合は、この酵素のT形態からR形態への変化に相当し、サブユニット境界での3次構造を少し変化させ、それにより4次構造を大きく変化させる。[7] AMPの結合は2つのサブユニットのタワーヘリックス(262-278残基)を50度回転させ、構造とサブユニット間の相互作用を大きく変える。このタワーヘリックスの回転により、2つのサブユニットを10度回転させ、重要なことに、T形態では触媒部位への接触を妨げていた282-286残基(280番台のループ)を不規則にし、R形態では妨げられなくする。[5]
最後に述べる、おそらくグリコーゲンホスホリラーゼタンパク質で最も興味深い部位は、グリコーゲン貯蔵部位と呼ばれる。397-437残基がこの構造を形成し、このタンパク質がその触媒部位から全長30 Åにわたってグリコーゲン鎖と共有結合ができる。この部位は末端グルコース分子の分解を始める前にこの酵素が結合している部位である可能性が最も高い。事実、細胞に存在する70%のホスホリラーゼ2量体がグリコーゲン顆粒に結合している。[8]
哺乳類では、グリコーゲンホスホリラーゼの主要なアイソザイムは筋、肝、および脳でみられる。脳のタイプは成体の脳と胚性組織で優勢であり、肝および筋のタイプは成体の肝と骨格筋でそれぞれ優勢である。[9]
臨床での重要性
[編集]| グリコーゲンホスホリラーゼ;筋 (マッカードル病、糖原病V型) | |
|---|---|
| 識別子 | |
| 略号 | PYGM |
| Entrez | 5837 |
| HUGO | 9726 |
| OMIM | 608455 |
| RefSeq | NM_005609 |
| UniProt | P11217 |
| 他のデータ | |
| EC番号 (KEGG) | 2.4.1.1 |
| 遺伝子座 | Chr. 11 q12-q13.2 |
| グリコーゲンホスホリラーゼ;肝(エール病、糖原病VI型) | |
|---|---|
| 識別子 | |
| 略号 | PYGL |
| Entrez | 5836 |
| HUGO | 9725 |
| OMIM | 232700 |
| RefSeq | NM_002863 |
| UniProt | P06737 |
| 他のデータ | |
| EC番号 (KEGG) | 2.4.1.1 |
| 遺伝子座 | Chr. 14 q11.2-24.3 |
| グリコーゲンホスホリラーゼ;脳 | |
|---|---|
| 識別子 | |
| 略号 | PYGB |
| Entrez | 5834 |
| HUGO | 9723 |
| OMIM | 138550 |
| RefSeq | NM_002862 |
| UniProt | P11216 |
| 他のデータ | |
| EC番号 (KEGG) | 2.4.1.1 |
| 遺伝子座 | Chr. 20 p11.2-p11.1 |
グリコーゲンホスホリラーゼの阻害は2型糖尿病の治療の一つの方法として提唱されてきた。[10] 肝でのグルコース産生は2型糖尿病の患者で増加することが示されてきたため、[11] 肝に貯蔵されたグリコーゲンからのグルコース放出を阻害することは確かな方法に見える。ヒト肝のグリコーゲンホスホリラーゼ(HLGP)のクローニングにより、研究で普段使われていたウサギ筋グリコーゲンホスホリラーゼ(RMGP)には存在しないサブユニット境界付近のアロステリック部位がその酵素で新規に見つかった。この部位はAMPアロステリック部位での阻害剤と同じものには感受性ではなく、[12] そして、最も大きな成功はグルコースの構造の類似体である新しい阻害剤を合成することができたことである。それ以来、グルコース-6-リン酸は有名なHLPG阻害剤であり、比較的活性が低いT状態を安定化させる。[13] これらのグルコース派生物はHLPGの阻害にいくらか成功をおさめ、予測されたKi値は0.016 mMと低い。[14]
グリコーゲンホスホリラーゼの筋のアイソフォーム(PYGM)の変異はマッカードル病 (糖原病V型)と関係する。今日までに65を超える、マッカードル病を引き起こすPYGM遺伝子での変異が特定されてきた。[15][16] マッカードル病の症状は筋委縮、筋痛症、および持久力の低下であり、骨格筋でのグルコース濃度が低いことに起因する。[17]
グリコーゲンホスホリラーゼの肝のアイソフォーム(PYGL)の変異は、エール病 (糖原病VI型)と関係する。[18][19] エール病は通常低血糖に限られる軽い症状を示すが、残存酵素活性のために診断が難しいことがある。[20]
グリコーゲンホスホリラーゼの脳のアイソフォーム(PYGLB)は胃がんのバイオマーカーとして提唱されている。[21]
調節
[編集]グリコーゲンホスホリラーゼは、アロステリックな調節とリン酸化の両方で調節されている。.
アドレナリン、インスリンおよびグルカゴンのようなホルモンは、Gタンパク質と関連した情報伝達物質による信号増幅系を用いてグリコーゲンホスホリラーゼを調節する。アドレナリンはヘテロ3量体Gタンパク質と共役した7回膜貫通受容体を通してアデニル酸環化酵素を活性化し、細胞内のサイクリックAMPの濃度を上昇させる。サイクリックAMPはタンパク質キナーゼA(PKA)と結合し、活性化状態のものを遊離させる。次にPKAはホスホリラーゼキナーゼをリン酸化し、そしてそれはグリコーゲンホスホリラーゼbをリン酸化し、活性型のグリコーゲンホスホリラーゼaに変える。このリン酸化はグリコーゲンホスホリラーゼbの14番目のセリンのリン酸化である。肝では、グルカゴンが他の連鎖反応を引き起こすもうひとつのGタンパク質結合受容体を活性化し、その結果ホスホリパーゼC(PLC)を活性化させる。PLCは間接的に肝細胞の小胞体から細胞質へカルシウムイオンを放出させる。カルシウムイオンはカルモジュリンサブユニットに結合し、グリコーゲンホスホリラーゼキナーゼを活性化させる。グリコーゲンホスホリラーゼキナーゼはすでに述べたような仕方でグリコーゲンホスホリラーゼを活性化する。
グリコーゲンホスホリラーゼbは筋では常に不活性であるのではなく、AMPによってアロステリックに活性化されることがある。激しい運動により上昇したAMP濃度は、エネルギーを要求する信号となる。AMPはグリコーゲンホスホリラーゼb の構造を緊張型(T)から弛緩型(R)に変えることで活性化する。この弛緩型はリン酸化された酵素と似た性質を持つ。ATP濃度の上昇は十分なエネルギー貯蔵を意味し、AMPをヌクレオチド結合部位から除き、この種の活性化を阻害する。
食事を摂るとインスリンが分泌され、血中のグルコース濃度の上昇の信号となる。インスリンは間接的にPP-1とホスホジエステラーゼを活性化する。PP-1は直接グリコーゲンホスホリラーゼaを脱リン酸化し、不活性なグリコーゲンホスホリラーゼbに戻す。ホスホジエステラーゼはサイクリックAMPをAMPにする。この活性は(グルカゴンとアドレナリンによって増幅した)情報伝達物質を除き、PKAを阻害する。こうなると、PKAは(活性型)グリコーゲンホスホリラーゼaにするリン酸化連鎖反応をもはや引き起こせない。インスリンによって始まったこれらの作用により、グリコーゲンの分解は終わり、グリコーゲンの産生が始まる。
ホスホリラーゼaとホスホリラーゼbはそれぞれT(緊張)不活性状態と R(弛緩)状態で存在する。ホスホリラーゼbは通常T状態であり、ATPとグルコース-6-リン酸の存在により不活性である。ホスホリラーゼaは通常R 状態(活性型)である。
肝のグリコーゲンホスホリラーゼのアイソザイムはグルコース濃度感受性であり、それは肝がグルコースを供給する器官だからである。本質的には、肝のホスホリラーゼはグルコースに反応し、R状態をT状態にすぐに変換し、不活性化する。さらに肝のホスホリラーゼはAMP非感受性である。
歴史的重要性
[編集]グリコーゲンホスホリラーゼはアロステリックな酵素として発見された最初の酵素である。[7] この発見はCarlとGerty Coriによってなされた多くの業績のうちのひとつである。1943年、Arda Greenの助けを借り、その夫婦はグリコーゲンホスホリラーゼがそのリン酸化状態によってaまたはbの形態をとり、AMPの有無によりRまたはTの形態をとることを説明した。[22]
脚注
[編集]- ^ PDB: 3E3N
- ^ a b Livanova NB, Chebotareva NA, Eronina TB, Kurganov BI (May 2002), “Pyridoxal 5′_Phosphate as a Catalytic and Conformational Cofactor of Muscle Glycogen Phosphorylase b”, Biochemistry (Moscow) 67 (10): 1089–1998, doi:10.1023/A:1020978825802, PMID 12460107
- ^ Palm D, Klein HW, Schinzel R, Buehner M, Helmreich, EJM (February 1990), “The role of pyridoxal 5'-phosphate in glycogen phosphorylase catalysis”, Biochemistry 29 (5): 1099–1107, doi:10.1021/bi00457a001, PMID 2182117
- ^ Browner MF, Fletterick RJ (February 1992), “Phosphorylase: a biological transducer”, Trends in Biochemical Science 17 (2): 66–71, doi:10.1016/0968-0004(92)90504-3, PMID 1566331
- ^ a b Johnson LN (March 1992), “Glycogen phosphorylase: control by phosphorylation and allosteric effectors”, FASEB Journal 6 (6): 2274–82, PMID 1544539
- ^ Newgard CB, Hwang PK, Fletterick, RJ (1989), “The family of glycogen phosphorylases: structure and function”, Critical Reviews Biochemistry and Molecular Biology 24 (1): 69–99, doi:10.3109/10409238909082552, PMID 2667896
- ^ a b Johnson LN, Barford, D (February 1990), “Glycogen phosphorylase. The structural basis of the allosteric response and comparison with other allosteric proteins.”, Journal of Biological Chemistry 265 (5): 2409–2412, PMID 2137445
- ^ Meyer F, Heilmeyer LM Jr, Haschke RH, Fischer EH (Dec 1970), “Control of phosphorylase activity in a muscle glycogen particle. I. Isolation and characterization of the protein-glycogen complex”, Journal of Biological Chemistry 245 (24): 6642–6648, PMID 4320610
- ^ David ES, Crerar MM (January 1986), “Quantitation of muscle glycogen phosphorylase mRNA and enzyme amounts in adult rat tissues”, Biochim. Biophys. Acta 880 (1): 78–90, PMID 3510670
- ^ Somsák L, Nagya V, Hadady Z, Docsa T, Gergely P. (2003), “Glucose analog inhibitors of glycogen phosphorylases as potential antidiabetic agents: recent developments”, Current Pharmacological Design 9 (15): 1177–89, doi:10.2174/1381612033454919, PMID 12769745
- ^ Moller DE (Dec 2001), “New drug targets for type 2 diabetes and the metabolic syndrome”, Nature 414 (6865): 821–7, doi:10.1038/414821a, PMID 11742415
- ^ Coats WS, Browner MF, Fletterick RJ, Newgard CB (Aug 1991), “An engineered liver glycogen phosphorylase with AMP allosteric activation”, Journal of Biological Chemistry 266 (24): 16113–9, PMID 1874749
- ^ Oikonomakos NG, Kontou M, Zographos SE, Tsitoura HS, Johnson LN, Watson KA, Mitchell EP, Fleet GW, Son JC, Bichard CJ, et al. (Jul 1994), “The design of potential antidiabetic drugs: experimental investigation of a number of beta-D-glucose analogue inhibitors of glycogen phosphorylase”, European Journal of Drug Metabolism and Pharmacology 19 (3): 185–92, doi:10.1007/BF03188920, PMID 7867660
- ^ Hopfinger A J, Reaka A, Venkatarangan P, Duca J S, Wang S. (Sep 1999), “Prediction of Ligand−Receptor Binding Free Energy by 4D-QSAR Analysis: Application to a Set of Glucose Analogue Inhibitors of Glycogen Phosphorylase”, Journal of Chemical Information and Computer Science 39: 1141–1150, doi:10.1021/ci9900332
- ^ Nogales-Gadea G, Arenas J, Andreu AL (January 2007), “Molecular genetics of McArdle's disease”, Curr Neurol Neurosci Rep 7 (1): 84–92, doi:10.1007/s11910-007-0026-2, PMID 17217859
- ^ Andreu AL, Nogales-Gadea G, Cassandrini D, Arenas J, Bruno C (July 2007), “McArdle disease: molecular genetic update”, Acta Myol 26 (1): 53–7, PMID 17915571
- ^ Grünfeld JP, Ganeval D, Chanard J, Fardeau M, Dreyfus JC (Jun 1972), “Acute renal failure in McArdle's disease. Report of two cases”, New England Journal of Medicine 286 (23): 1237–41, doi:10.1056/NEJM197206082862304, PMID 4502558
- ^ Burwinkel B, Bakker HD, Herschkovitz E, Moses SW, Shin YS, Kilimann MW (April 1998), “Mutations in the liver glycogen phosphorylase gene (PYGL) underlying glycogenosis type VI”, Am. J. Hum. Genet. 62 (4): 785–91, doi:10.1086/301790, PMC 1377030, PMID 9529348
- ^ Chang S, Rosenberg MJ, Morton H, Francomano CA, Biesecker LG (May 1998), “Identification of a mutation in liver glycogen phosphorylase in glycogen storage disease type VI”, Hum. Mol. Genet. 7 (5): 865–70, doi:10.1093/hmg/7.5.865, PMID 9536091
- ^ Tang NL, Hui J, Young E, Worthington V, To KF, Cheung KL, Li CK, Fok TF (Jun 2003), “A novel mutation (G233D) in the glycogen phosphorylase gene in a patient with hepatic glycogen storage disease and residual enzyme activity”, Molecular Genetics and Metabolism 79 (2): 142–145, doi:10.1016/S1096-7192(03)00068-4, PMID 12809646
- ^ Shimada S, Matsuzaki H, Marutsuka T, Shiomori K, Ogawa M (July 2001), “Gastric and intestinal phenotypes of gastric carcinoma with reference to expression of brain (fetal)-type glycogen phosphorylase”, J. Gastroenterol. 36 (7): 457–64, doi:10.1007/s005350170068, PMID 11480789
- ^ Cori GT, Green AA (July 1943), “Crystalline muscle phosphorylase II prosthetic group”, Journal of Biological Chemistry 151 (1): 21–29
参考文献
[編集]- Voet, Judith G.; Voet, Donald (1995), “Chapter 17: Glycogen Metabolism”, Biochemistry (2nd ed.), New York: J. Wiley & Sons, ISBN 0-471-58651-X
- Voet, Judith G.; Voet, Donald (2004), “Chapter 18: Glycogen Metabolism”, Biochemistry (3rd ed.), New York: J. Wiley & Sons, ISBN 0-471-19350-X
外部リンク
[編集]- GeneReviews/NCBI/NIH/UW entry on Glycogen Storage Disease Type VI - Hers disease
- Glycogen phosphorylase - MeSH・アメリカ国立医学図書館・生命科学用語シソーラス
- Diwan JJ. “Glycogen Metabolism”. Molecular Biochemistry I. Rensselaer Polytechnic Institute. 2009年1月10日閲覧。
- Goodsell DS (2001年12月1日). “Glycogen Phosphorylase”. Molecule of the Month. RCSB Protein Data Bank. 2009年1月10日閲覧。


